作者:谭高翼 摄影:谭高翼
3月22日,国际著名学术期刊Nucleic Acids Research (《核酸研究》,IF=16.971)在线发表我校生物反应器工程国家重点实验室和生物工程学院在放线菌大型基因簇捕捉和沉默基因簇激活发掘新颖活性天然产物研究中取得的最新进展。研究论文标题为“Activating crypticbiosynthetic gene cluster through a CRISPR-Cas12a mediated direct cloningapproach”。
图片说明:学术期刊Nucleic Acid Research在线发表放线菌基因簇捕捉和沉默基因激活新进展
放线菌是合成天然产物药物的主力,被广泛用于抗肿瘤药物(如多柔比星)、免疫抑制剂(如FK506)和杀虫剂(如阿维菌素)等多种药物的发酵和规模化工业制造。然而,放线菌基因组DNA的GC含量大多超过70%,其中的大型沉默天然产物编码基因簇的编辑/克隆和激活表达,一直是限制放线菌活性天然产物发掘与合成的技术难点。聚焦这一难题,该研究利用CRISPR-Cas12a的精准靶向切割特性和琼脂糖凝胶包埋制备高质量基因组DNA的技术,成功开发了一种从放线菌基因组中高效克隆/编辑大片段DNA的方法,简称CAT-FISHING(CRISPR-Cas12a-mediated fast direct biosynthetic gene cluster cloning, 猫捕),将其用于放线菌大型沉默基因簇的克隆和激活表达,最终实现活性新颖天然产物的发掘。
这项研究不仅提供了一种快速、高效的大型DNA片段编辑/克隆策略,通过体外直接克隆,将放线菌基因簇的克隆上限突破至145kb,实验成本相较常规建库的方法降低了125倍。对批量克隆得到的基因簇进行激活表达获得一种结构新颖的大环内酰胺类化合物,命名为marinolactam A(马里诺霉素A)。在随后的活性测试中,研究发现该化合物对结肠癌细胞HCT116具有较好的杀灭活性(IC50= 18 μg/mL)。上述研究结果表明,CAT-FISHING作为一种高效的大型基因簇克隆的平台技术,将极大促进放线菌天然产物的发掘和新颖活性药物发现。
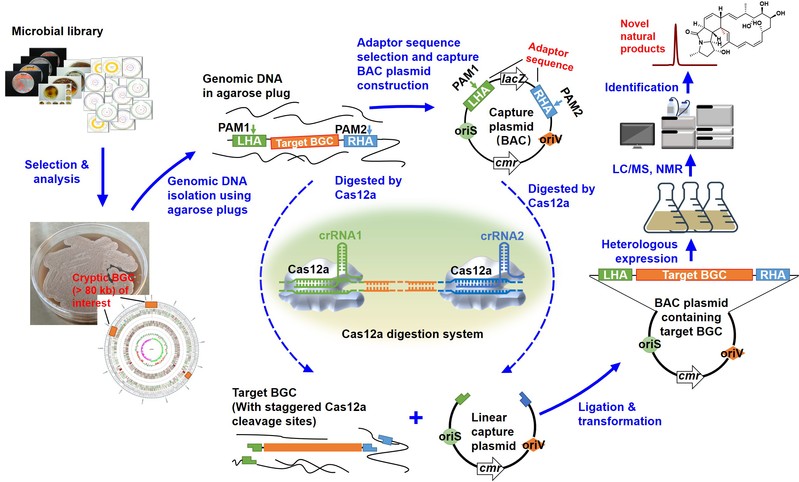
图片说明:基于CAT-FISHING的大型基因簇捕捉和新颖活性天然产物发掘
这项研究自bioRxiv预印版刊登以来,受到领域内同行专家的认可和好评,例如主流期刊Current Opinion inBiotechnology亮点评述这项成果,认为这是迄今最快捷的大型基因簇克隆技术(“CAT-FISHINGprovides one of the fastest cloning strategy for the large BGC so far”)。该技术已经申请专利(专利号:202010575747.5;国际申请号:PCT/CN2020/120332)。
我校博士生梁敏东、曾晓倩和硕士生刘乐诗,以及浙江大学的徐飞研究员为本文共同第一作者。我校生物反应器工程国家重点实验室张立新教授团队的谭高翼副研究员为本文通讯作者。我校生物反应器工程国家重点实验室和生物工程学院为本文唯一通讯单位。该工作得到了国家重点研发计划、国家自然科学基金等多个项目的支持。
据悉,在生物反应器工程国家重点实验室主任张立新教授的领导下,谭高翼副研究员和团队成员自2018年以来一直围绕CRISPR/Cas12a进行持续攻关和研发,迄今已经开发并形成多项“CAT”系列的使能生物技术。包括:能实现微生物代谢物小分子(例如天然产物,活性小分子等)高通量、快速筛选和检测的CAT-SMELOR平台(Nature Communications, 2019);以及随后升级得到的CAT-SMELOR 2.0平台(Science Bulletin, 2020),这不仅可向下兼容小分子检测,同时可“即插即用”实现对生物大分子(例如蛋白质、酶等)的高效检测;还有最近开发的CAT-SMELOR-Covid技术(Synthetic and SystemBiotechnology, 2021),进一步将该检测平台升级扩展到了包括病毒抗原检测在内的医学诊断领域;本报道中的CAT-FISHING技术(NucleicAcid Research, 2022)通过对大型基因簇的克隆和激活表达,打通了活性新颖代谢物小分子发掘的关键技术瓶颈。这些成果聚焦生物医药和健康领域,为微生物活性分子的发掘、检测以及后续的高效生物制备体系开发提供了全链条的技术支撑体系,对于微生物天然产物药物的研发和制造具有重要的意义。
论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkac181/6552061?login=true