作者:马培强
华东理工大学生物反应器工程国家重点实验室叶邦策教授课题组通过模块化设计DNA分子机器,开发了一种多DNA机器交互网络系统,成功模拟了脊椎动物获得性免疫系统中细胞与细胞间通讯对病原体的识别,清除以及记忆。通过三种DNA分子机器分别模拟关键免疫细胞:树突状细胞(DC)、T细胞和B细胞,生动地展示了病原体侵袭过程中DC细胞,T淋巴细胞和B淋巴细胞的连续激活、抗体分泌、记忆形成和快速高效的二次应答。
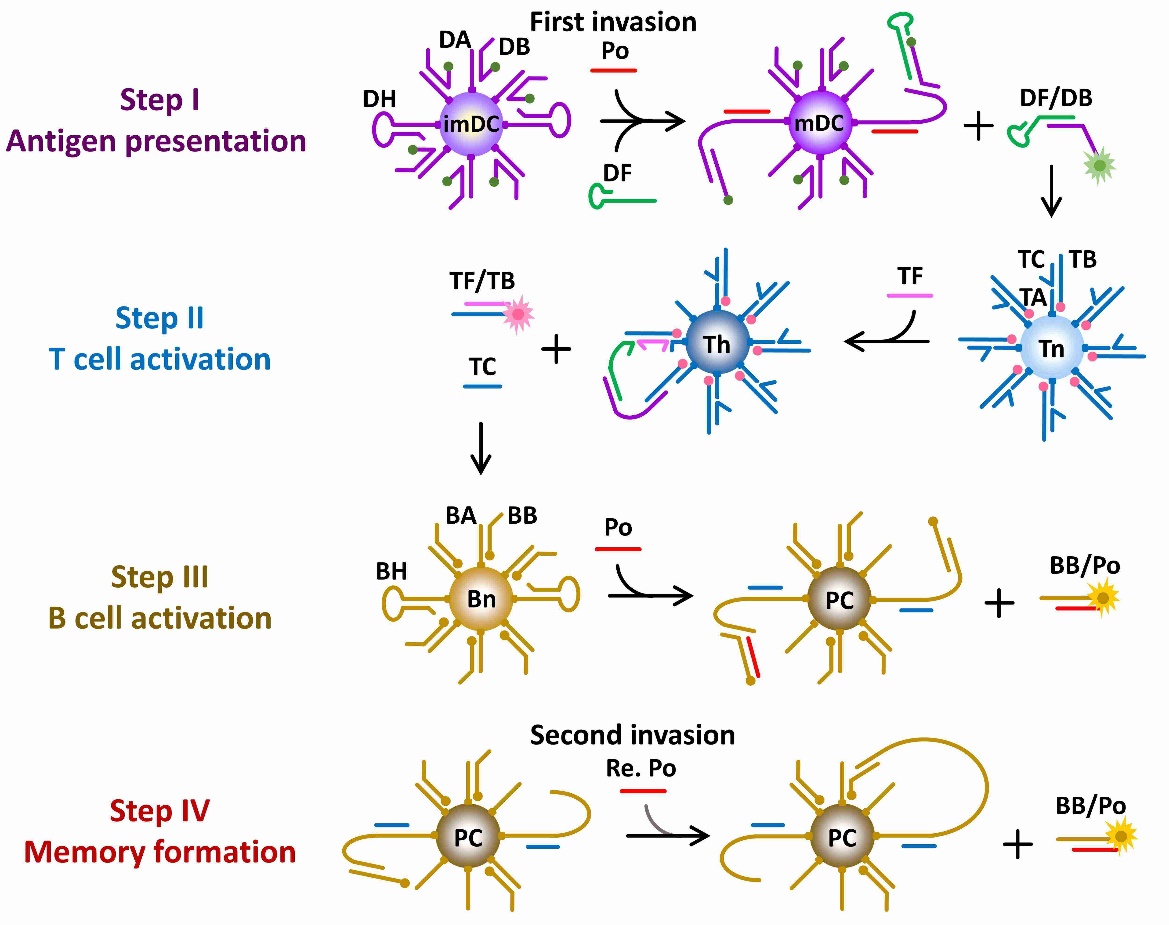
图片说明:多机器交互网络系统模拟适应性免疫应答反应的原理示意图。
利用一段HIV病毒基因序列模拟病毒侵入过程,当HIV序列不存在时,该系统中三种DNA分子机器均处于稳定的初始状态。当HIV序列首次入侵后,机器DC由未成熟状态变为成熟状态,将抗原呈递给机器T,使其由初始态转变为辅助T细胞状态,进而激活机器B释放模拟抗体链与病原体结合,模拟免疫系统首次应答过程。当病原体再次入侵上述系统,无需机器DC和机器T参与,第一次应答过程中由机器B转变成的机器PC可直接释放抗体链与病原体结合,该过程比初次应答反应更高效,进而模拟了免疫系统的记忆功能及二次应答。模块化设计可将该网络交互系统进一步升级,响应于不同的分子,如肿瘤标志物miRNA或蛋白、释放小干扰RNA、反义寡核苷酸或miRNA抑制剂等,有望拓展开发感知细胞内多种刺激物的智能靶向诊疗系统。该工作将为DNA分子机器多样化设计及高级生物应用(如疾病早期诊断治疗、药物智能载运和释放等)提供全新的研究思路。英国皇家化学会Chemistry World并对此工作进行了详细评述(https://www.chemistryworld.com/news/first-three-component-artificial-immune-system-made-of-dna-studded-gold-nanomachines/4011282.article)。
该研究工作由博士生马培强、硕士生黄庆和李华栋完成,以“A Multimachine Communication Network that Mimics the Adaptive Immune Response”为题发表在国际著名学术期刊《美国化学会志》,尹斌成教授和叶邦策教授为文章的共同通讯作者。该研究得到了国家自然科学基金委优秀青年基金项目、重点基金等项目资助。
原文链接: https://pubs.acs.org/doi/abs/10.1021/jacs.9b11545